Survival Analysis (2/3)
지난 포스트에서는 Survival Analysis를 간략하게 설명하고, Survival function과 Cumulative hazard function을 추정하는 방법과 두 그룹의 생존 양상을 비교하는 Logrank test를 소개했습니다. 이번 포스트에서는 각 대상이 사망할지(즉, 이탈할지) 예측하는 여러 방법을 소개합니다.
물론 지난 포스트에서 설명한 Kaplan-Meier estimation도 생존과 이탈을 예측하는 가장 단순한 방법입니다. 예를 들어 지금까지 신규 가입 고객 10억 명 중 20%가 한 달 내 이탈하는 양상을 보였다면, 오늘 가입한 천 만명 중 20%는 한 달 내 이탈할 것이라 단순히 예측할 수 있습니다. 하지만 이 예측 결과는 각 대상의 데이터, 즉 feature를 전혀 고려하지 않았습니다. 하지만 feature별로 이탈 확률이 다르다면 더 나은 예측을 할 수 있겠죠.
Survival Prediction 관련 연구는 다음 그림[1]과 같이 크게 네 가지로 나뉩니다.
- Non-parametric: 데이터의 feature와 생존 시간 분포 정보를 사용하지 않는 방법입니다. 분포 정보를 알 수 없을 때 유용하지만 예측이 부정확할 수 있습니다.
- Semi-parametric: feature 정보를 활용하지만, 생존 시간 분포 정보를 사용하지 않는 방법입니다. Cox Proportional Hazard 모델이 해당합니다.
- Parametric: 생존 시간 분포가 존재한다고 가정하고 회귀 모델로 생존 시간을 예측하는 기법입니다. 지수 분포, 베이불(Weibull) 분포, 로지스틱(Logistic) 분포, 정규 분포 등을 사용합니다.
- Machine learning: 다양한 머신러닝 알고리즘을 생존 분석에 적용한 연구 결과들입니다.
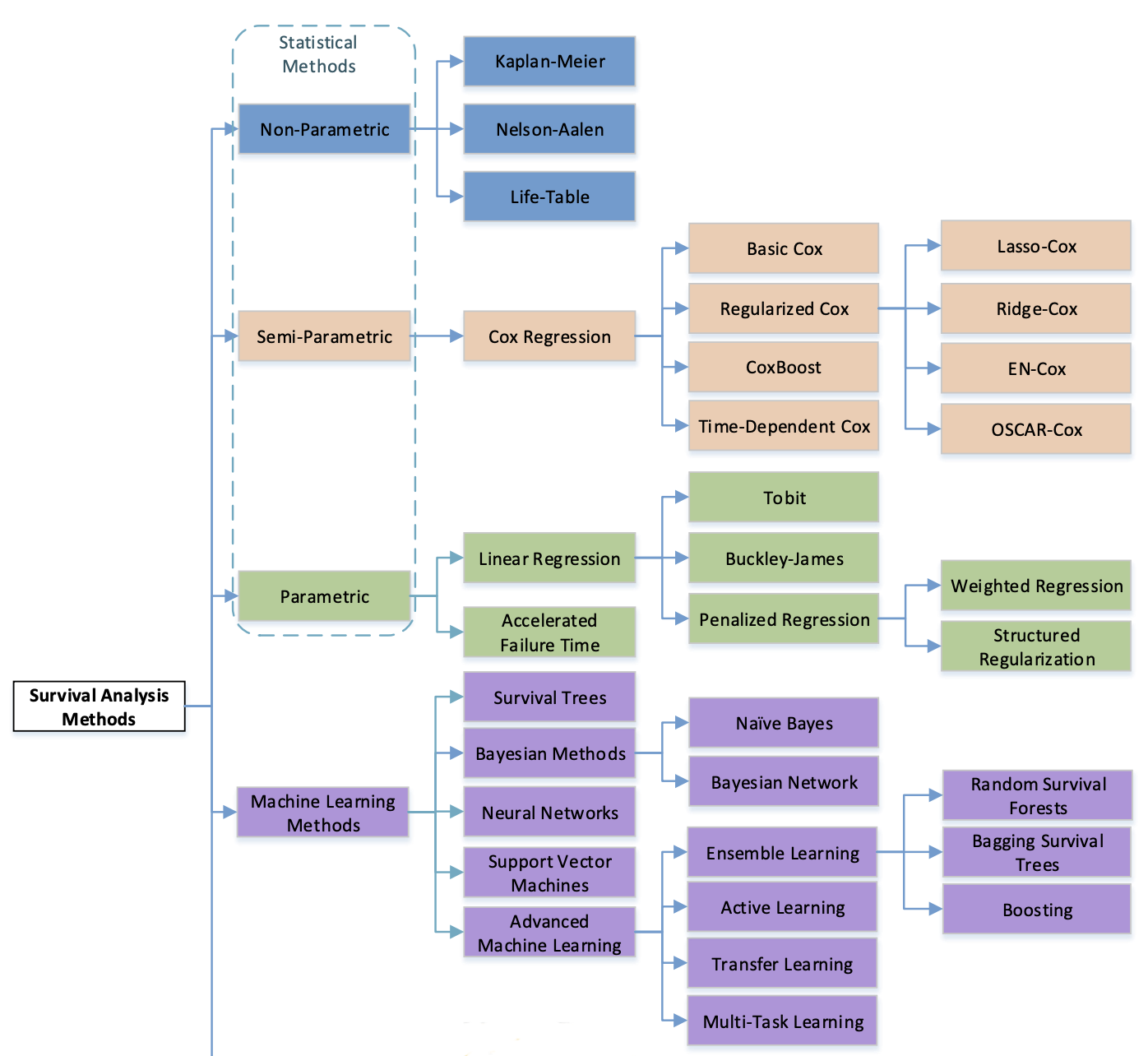 그림 1. Survival Analysis 기법 분류 [1]
그림 1. Survival Analysis 기법 분류 [1]
이번 포스트에서는 그림 1의 여러 기법 중 (Basic) Cox Proportional Hazard (Cox PH), Weibull Accelerated Failure Time (Weibull AFT), Support Vector Machine (SVM), Gradient Boosting 기법을 조금 더 자세히 설명하고, 파이썬으로 어떻게 구현하는지 소개하겠습니다. 각 기법의 이론적인 내용은 다음 포스트에서 자세히 다룹니다.
예제 데이터 준비
이번 포스트에서는 예제 데이터가 훨씬 더 많이 필요하고 Feature도 만들어야 합니다. 다음과 같이 Feature를 정의하고 무작위로 생성하겠습니다.
예제 데이터 생성 규칙
| Feature | 값 생성 분포 | 생존 확률 계산 | 가중치 |
|---|---|---|---|
| gender (성별) | 범주, 균등분포 (남성=0, 여성=1) | 각 성별 정규분포 | 0.3 |
| age (나이) | 정수, 감마분포 (\(k=15, \theta=1.6\)) | 연령별 정규분포 - log(시간) * 0.02 | 0.35 |
| num_friends (친구 수) | 정수, 감마분포 (\(k=3, \theta=10\)) | log(값) / 5 | 0.05 |
| num_liked (좋아요 받은 수) | 정수, 감마분포 (\(k=5, \theta=30\)) | log(값) / 6 | 0.05 |
| num_calls (통화 수) | 정수, 친구 수 * 정규분포 (\(\mu=15, \sigma=2\)) | log(값) / 8 + log(시간) * 0.03 | 0.25 |
각 고객의 생존 확률은 위 Feature로 계산한 생존 확률에 가중치를 곱해 계산합니다. 그런 다음, 매 시각 생존 확률이 70% 미만인 고객을 해당 시각에 이탈했다고 가정합니다. ‘나이’와 ‘통화 수’ 변수에 시간 변수를 추가한 부분을 눈여겨 보세요. 이 시간 변수의 효과는 다음 포스트에서 Cox PH 모델을 설명할 때 다시 언급하겠습니다.
예제 데이터를 생성하는 코드는 다음과 같습니다.
import numpy as np
import pandas as pd
def generate_features(num_samples):
genders = np.random.randint(0, 2, num_samples)
ages = np.random.gamma(15, 1.6, num_samples).round()
nums_friends = np.random.gamma(3, 10, num_samples).round()
nums_liked = np.random.gamma(5, 30, num_samples).round()
nums_calls = np.multiply(nums_friends, np.random.normal(15, 2, num_samples).clip(1)).round()
return pd.DataFrame({
'gender': genders,
'age': ages,
'num_friends': nums_friends,
'num_liked': nums_liked,
'num_calls': nums_calls
})
def get_normal_prob(mean, stdev):
return np.clip(np.random.normal(mean, stdev), 0.1, 0.99)
def get_death_prob(t, features):
zipped = pd.DataFrame({
'gender': features['gender'].apply(
lambda x:
get_normal_prob(0.9, 0.01) if x == 0
else get_normal_prob(0.85, 0.01)
),
'age' : features['age'].apply(
lambda x: ((0 <= x < 10 and get_normal_prob(0.7, 0.02)) or
(10 <= x < 20 and get_normal_prob(0.85, 0.03)) or
(20 <= x < 30 and get_normal_prob(0.9, 0.05)) or
(30 <= x < 40 and get_normal_prob(0.75, 0.05)) or
(40 <= x < 50 and get_normal_prob(0.7, 0.05)) or
get_normal_prob(0.2, 0.05)) - np.random.normal(0.02 * np.log(t), 0.005)
),
'num_friends': features['num_friends'].apply(
lambda x: np.clip(np.log(max(1, x)) * 0.2, 0.1, 0.99)
),
'num_liked': features['num_liked'].apply(
lambda x: np.clip(np.log(max(1, x)) * (1/6), 0.1, 0.99)
),
'num_calls': features['num_calls'].apply(
lambda x: np.clip(np.log(max(1, x)) * (1/8) + np.random.normal(0.03 * np.log(t), 0.005), 0.1, 0.99)
)
})
return zipped['gender'] * 0.3 + zipped['age'] * 0.35 + zipped['num_friends'] * 0.05 + zipped['num_liked'] * 0.05 + zipped['num_calls'] * 0.25
def generate_dataset(num_samples, death_threshold, max_time):
data = generate_features(num_samples)
data['event'] = False
data['time'] = 1
time = 1
while time < max_time:
time = time + 1
prob = get_death_prob(time, data)
data['time'] = data['time'] + ~(data['event'] | (prob < death_threshold)) * 1
data['event'] = data['event'] | (prob < death_threshold)
return data
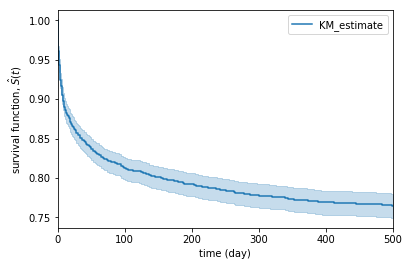
이제 무작위로 3,000명을 생성하고 매일 생존 확률을 계산해 확률이 75% 미만인 고객을 이탈시킵니다. 500일까지 생존한 고객은 이탈하지 않았다고 간주합니다. survival function을 그려보면 500일 동안 대략 25%가 점차 이탈하는 것을 볼 수 있습니다.
from lifelines import KaplanMeierFitter
data = generate_dataset(3000, 0.75, 500)
kmf = KaplanMeierFitter()
kmf.fit(data["time"], data["event"])
plot = kmf.plot_survival_function()
plot.set_xlabel('time (day)')
plot.set_ylabel('survival function, $\hat{S}(t)$')
plot
데이터 준비를 마쳤습니다. 이제 이 데이터로 여러 예측 모델을 만들어 보겠습니다.
Semi-parametric methods
Basic Cox Proportional Hazard model
Cox Proportional Hazard (Cox PH) 모델은 위험함수(hazard function)를 기저위험(underlying baseline hazard)와 매개변수로 나누어 표현하는 기법입니다. 다시 말해, 위험함수를 다음 공식으로 정의합니다.
\[\lambda(t|X_i) = \lambda_0(t) \exp(\beta_1 X_{i1} + \cdots + \beta_p X_{ip}) = \lambda_0(t) \exp(X_i \cdot \beta)\]Cox PH의 이론적인 내용은 다음 포스트에서 더 자세히 다루겠습니다. 지금은 시간 \(t\)가 기저위험함수에만 있다는 점을 눈여겨 보세요. 다시 말해, Basic Cox PH 모델은 매개변수가 시간과 무관하게 생존에 영향을 준다고 가정합니다. (물론 그렇게 좋은 가정이 아닐 겁니다. 하지만 시간의 효과를 무시해도 괜찮을 때도 많죠.)
이제 Cox PH 모델을 만들어 봅시다.
from lifelines import CoxPHFitter
from lifelines.utils import k_fold_cross_validation
cox = CoxPHFitter()
#fitting
cox.fit(data, duration_col='time', event_col='event', show_progress=True)
cox.print_summary()
cox.plot()
#cross-validation
cox_cv_result = k_fold_cross_validation(cox, data, duration_col='time', event_col='event', k=5)
print('C-index(cross-validation) = ', np.mean(cox_cv_result))
데이터를 무작위로 생성했기 때문에 결과는 조금씩 달라지겠지만, 교차 검증 결과는 대략 0.86~0.90 정도일 겁니다. 정확도는 Concordance Index(C-index)로 계산하며 0~1 사이의 값이고 높을수록 좋습니다. C-index 계산 방법과 그외 다른 정확도 지표 또한 다음 포스트에서 자세히 설명하겠습니다.
데이터가 나름 깔끔해서 그런지 예측 정확도가 높게 나왔습니다. 하지만 모델의 가설이 단순하기 때문에 최상의 결과라고 보기 어렵겠죠. 머신러닝 모델은 훨씬 더 좋은 결과를 낼 수 있습니다.
Parametric methods
Weibull Accelerated Failure Time model
다음으로 Parametric method를 살펴보겠습니다. 앞서 설명했듯이, 이 기법들은 생존 시간이 특정 분포를 따른다고 가정하고 회귀 기법을 사용해 생존 시간을 예측합니다.
가장 단순한 방법은 역시, noise term이 정규분포를 따른다고 가정하고 Linear Regression(선형회귀)을 적용하는 것입니다. 선형회귀를 활용한 생존 예측은 1958년에 처음 제안되었고[2], 이 밖에도 다양한 분포를 사용한 회귀 기법이 등장했습니다.
그 중 Weibull Accelerated Failure Time(Weibull AFT) 모델은 noise term이 Weibull(베이불) 분포를 따른다고 가정합니다. 또한 AFT라고 부르는 이유는, 각 변수가 생존 시간을 가속 또는 감속시킨다고 가정하기 때문입니다. 각 변수가 위험함수에 (시간과 무관한) 영향을 준다고 가정한 Cox PH 모델과는 조금 다릅니다. Weilbull AFT 모델이 생존 시간을 모델링하는 공식은 다음과 같습니다.
\[\log(t_i) = \beta_0 + \beta_1 x_{i1} + \cdots + \beta_p x_{ip} + \sigma \epsilon_i = \mathbf{X}_i\beta+ \sigma \epsilon_i\]공식은 선형회귀와 비슷하며, 단 noise term이 log-weibull 분포(즉, Gumble 분포)라는 점이 다릅니다. 따라서 선형회귀처럼 간단한 loss function(손실 함수)을 도출할 수 없고, 매우 복잡한 수학이 필요합니다. 관심이 있다면 관련 논문[3]을 참고하세요.
이제 Weibull AFT 모델을 만들어 봅시다. Cox PH와 마찬가지로 lifelines 패키지를 사용해 만들 수 있습니다.
from lifelines import WeibullAFTFitter
from lifelines.utils import k_fold_cross_validation
#fitting
aft = WeibullAFTFitter()
aft.fit(data, duration_col='time', event_col='event', show_progress=True)
aft.print_summary()
aft.plot()
#cross-validation
aft_cv_result = k_fold_cross_validation(aft, data, duration_col='time', event_col='event', k=5)
print('C-index(cross-validation) = ', np.mean(aft_cv_result))
Cox PH와 비슷하게, 0.87~0.90의 정확도가 나올 겁니다. 이제 머신러닝 모델로 넘어갑시다.
Machine Learning methods
Survival Support Vector Machine
Support Vector Machine(SVM)은 주로 classification(분류)에 사용하는 머신러닝 모델이지만, 회귀나 랭킹 문제에도 사용할 수 있는 모델도 제안되었습니다.
Survival SVM[4]은 이 랭킹 SVM을 확장해서, 예측 대상을 생존 시간에 따라 정렬하는 랭킹 모델을 학습하는 방식으로 생존을 예측합니다.
랭킹 SVM의 최적화 공식과 거의 동일하지만 censored(중도절단)를 고려한다는 점이 다릅니다.
중도절단된 대상은 기록된 생존 시간이 짧아도 다른 대상보다 일찍 사망(즉, 이탈)했다고 단정할 수 없기 때문에 정렬 대상에서 제외합니다.
파이썬 scikit-survival 패키지는 Survival SVM[4]과 Fast Survival SVM[5]을 각각 linear/kernel 버전으로 구현해 제공합니다. 여기서는 linear로 학습하는 방법을 소개하겠습니다.
from sksurv.svm import NaiveSurvivalSVM, FastSurvivalSVM
from sklearn.model_selection import GridSearchCV, KFold
X = data.drop(data.columns[-2:], axis=1)
y = data.apply(lambda x: (x.event, x.time), axis=1).to_numpy(dtype=[('event', 'bool'), ('time', 'float64')])
#SVM
svm = NaiveSurvivalSVM()
svm_params = {'alpha': np.arange(0.2, 1.5, 0.1)}
svm_gcv = GridSearchCV(svm, svm_params, cv=KFold(n_splits=5))
svm_result = svm_gcv.fit(X, y)
print('C-index = {}, (parameters: {})'.format(svm_result.best_score_, svm_result.best_params_))
#Fast SVM
fsvm = FastSurvivalSVM()
fsvm_params = {'alpha': np.arange(0.2, 1.5, 0.1), 'rank_ratio': np.arange(0, 1.1, 0.1)}
fsvm_gcv = GridSearchCV(fsvm, fsvm_params, cv=KFold(n_splits=5))
fsvm_result = fsvm_gcv.fit(X, y)
print('C-index = {}, (parameters: {})'.format(fsvm_result.best_score_, fsvm_result.best_params_))
정확도는 0.85~0.90 사이가 나옵니다. 앞서 만들었던 모델에 비해 크게 개선되지 않았습니다.
Gradient Boosting
scikit-survival 패키지는 Survival Gradient Boosted Regression도 제공합니다.
Cox PH 모델이 사용하는 partial likelihood loss를 기본 손실함수로 사용해 각 대상의 이탈(즉, 사망) 위험도를 예측합니다.
그 외 다른 손실함수(예: squared)를 지정하면 생존 시간을 예측합니다.
Cox PH를 사용해 만들어 봅시다. 아무 설정을 하지 않았지만, 0.96에 가까운 훨씬 더 좋은 정확도를 얻을 수 있습니다.
from sksurv.ensemble import GradientBoostingSurvivalAnalysis
gbr = GradientBoostingSurvivalAnalysis()
gbr_gcv = GridSearchCV(gbr, {}, cv=KFold(n_splits=5))
gbr_result = gbr_gcv.fit(X, y)
print('C-index = ', gbr_result.best_score_)
마무리
이번 포스트에서는 Survival Analysis 기법 중 생존을 예측하는 통계 & 머신러닝 기법들을 알아봤습니다. 고객 생존과 이탈을 사전에 예측할 수 있다면 실망한 고객이 떠나기 전에 다양한 조치를 취할 수 있을 겁니다. 그러나 이탈 예측 보다 더욱 중요한 것은 실제로 이탈을 방지하고 서비스 품질과 매출을 늘리는 일입니다. 이탈 예측의 정확도 기준도 고객에게 제안할 내용에 따라 달라질 수 있습니다.
이 포스트를 마지막으로 Survival Analysis에 대한 개괄적인 소개를 마칩니다. 마지막 세 번째 포스트는 지금까지 언급한 개념들의 이론적인 내용을 다룹니다.
References
[1] P. Wang, Y. Li, and C.K. Reddy, Machine Learning for Survival Analysis: A Survey, CoRR abs/1708.04649, 2017.
[2] J. Tobin, Estimation of Relationships for Limited Dependent Variables, Econometrica: Journal of the Econometric Society 26, 1, pp. 24-36, 1958.
[3] E. Liu, K. Lim, Using the Weibull Accelerated Failure Time Regression Model to Predict Time to Health Events, Rxivist, 2018.
[4] V. Van Belle, K. Pelckmans, J.A.K. Suykens, S. Van Huffel, Support Vector Machines for Survival Analysis, Int. Conf. on Computational Intelligence in Medicine and Healthcare, pp. 1-9, 2007
[5] S. Pölsterl, N. Navab, A. Katouzian, Fast Training of Support Vector Machines for Survival Analysis., Lecture Notes in Computer Science vol. 9285, 2015]
Read more
-
Survival Analysis (3/3)
Survival Analysis의 이론적 내용을 소개합니다. -
Survival Analysis (1/3)
Survival Analysis를 활용한 고객 이탈 분석 방법을 소개합니다.